
常见的蛋白互作研究方法有哪些呢?
本篇文章为大家整理了15种蛋白互作研究方法,快来看看吧~
一、融合蛋白沉降技术(Pull-down)
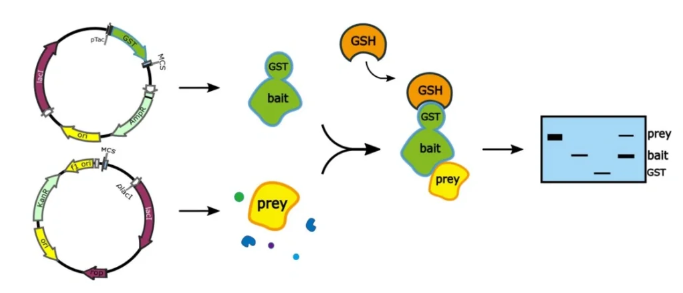
原理:通过磁珠/琼脂糖填料固定带有标记的“诱饵”蛋白,从细胞裂解液、纯化蛋白或表达系统中捕获“猎物”蛋白,然后将复合物蛋白分离后进行凝胶或质谱分析,从而确认互作蛋白的“身份”。
优点:不仅能证实靶蛋白的直接相互作用,且洗脱条件也很温和,保留蛋白质结构与天然蛋白复合体的同时,又避免将非特异性结合的蛋白质共同洗脱下来,确保了后续分析的准确性。
缺点:易出现假阳性;不能够完全反映细胞内蛋白真实互作的状态;融合表达的GST标签,肽链较长,可能会改变原目的蛋白的原有的折叠结构。
二、串联亲和纯化技术(TAP)
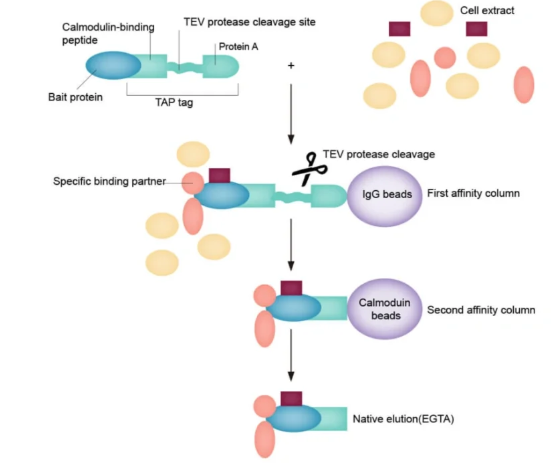
原理:传统的TAP标签蛋白由Protein A、TEV蛋白酶可剪切序列和钙调蛋白结合肽(CBP)组成。TAP技术通过两步亲和纯化来减少非特异性蛋白结合。
优点:TAP能减少非特异性蛋白结合,还能保留蛋白复合物在细胞内的修饰和结合状态。
缺点:需要较多的样本材料来提取蛋白,价格比较昂贵,不能高效检测到瞬时或较弱的蛋白质相互作用。
三、生物膜干涉技术(BLI)
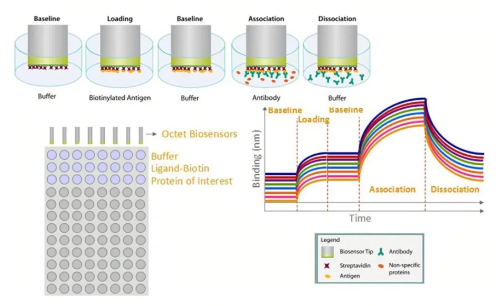
原理:是一种无标记(Label-free)光学生物传感技术,用于实时监测分析生物分子相互作用,并对其结合强度和动力学进行量化分析。与SPR技术不同的是,BLI技术中不使用金属芯片,而是利用生物传感器。将配体偶联在传感器的末端,浸入样品中来捕获分析物。
优点:易于使用;几乎无堵塞,适用于复杂、粘稠的样本;可最大限度地减小了体积效应。
缺点:传感器的灵敏度比SPR和GCI传感器低几个数量级;确定动力学参数时,精度有限。
四、表面等离子共振技术(SPR)
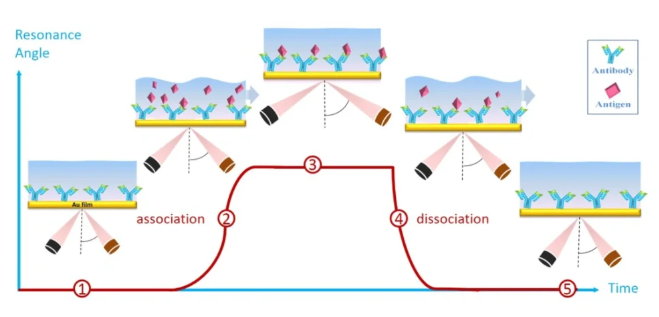
原理:在芯片表面固定一层生物分子识别膜,然后将待测样品流过芯片表面,若样品中有能够与芯片表面的生物分子识别膜相互作用的分子,会引起金膜表面折射率变化,最终导致SPR角变化,通过监测SPR的角度变化,获得被分析物的浓度、亲和力、动力学常数和特异性等。
优点:检测快捷方便,应用范围广、不需要标记待测物。
缺点:对于样品组成以及温度等干扰因素比较敏感,对于非特异性吸附比较难区分。
五、光栅耦合干涉技术(GCI)
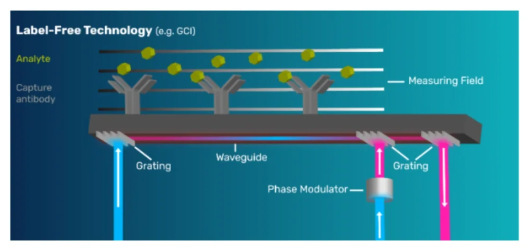
原理:基于波导干涉技术(另一种无标记的光学分析方法),可以实时监测和表征分子相互作用,并确定动力学速率参数、亲和常数以及与固定配体相互作用的分析物分子的浓度。
优点:初级灵敏度,适用于无标记的相互作用分析;无堵塞微流体;可以测量紧密结合剂和快速结合速率。
缺点:暂无
六、邻近蛋白标记技术(BioID)
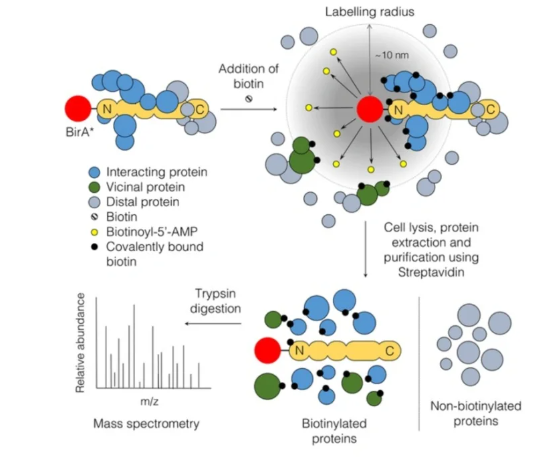
原理:通过将生物素连接酶(BirA)与“诱饵”蛋白融合表达,加入适量生物素,融合蛋白的相互作用以及附近环境中的蛋白质会被生物素化。然后通过链霉亲和素或Strep-Tactin进行亲和纯化,将被生物素化的蛋白质从细胞裂解液中富集出来,产物做质谱鉴定。
优点:BiolD鉴定到的互作蛋白是细胞内与诱饵蛋白天然互作的,符合体内真实生理情况,可信度高;弱结合蛋白和瞬时互作蛋白也能被检测到。
缺点:不能判断鉴定到的蛋白是直接还是间接与诱饵蛋白互作的,而没有伯胺的互作蛋白因不能被生物素化,所以无法被检测到。
七、酵母双杂交技术(Y2H)
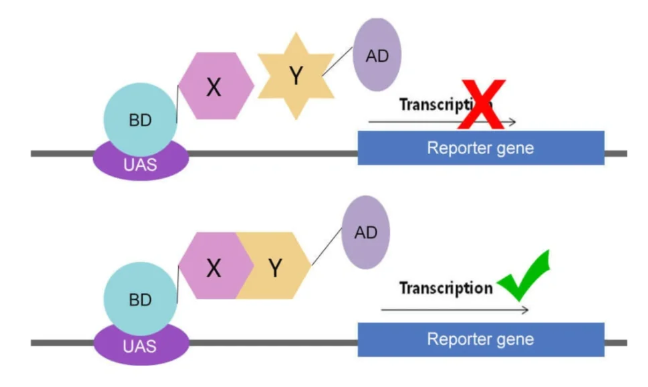
原理:很多真核生物的转录因子由两个有不同功能的结构域组成,包括转录激活结构域(Transcriptional activationdomain,AD)和DNA结合结构域(DNAbinding domain,BD)。转录因子通过BD和AD分别与DNA上的特异序列结合,从而启动相应基因的转录。
优点:简便、易用、费用低廉,能检测到瞬时或较弱的蛋白质相互作用。
缺点:较高的假阳性,不能真实反映蛋白质在自身细胞中的相互作用。
八、免疫共沉淀技术(Co-IP)
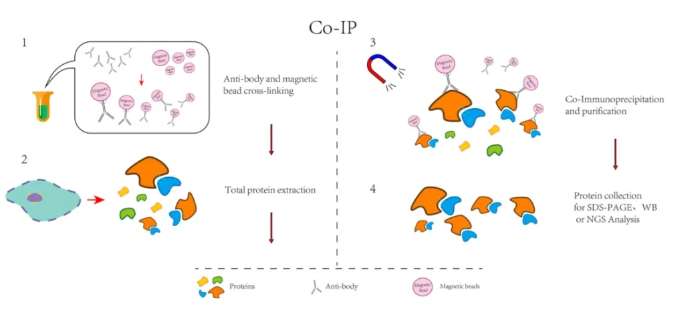
原理:以抗体和抗原之间的专一性作用为基础的用于研究蛋白质相互作用的经典方法,也是确定两种蛋白质在完整细胞内生理性相互作用的有效方法。当细胞在非变性条件下被裂解时,完整细胞内存在的许多蛋白质一蛋白质间的相互作用被保留了下来。如果用蛋白质X的抗体免疫沉淀X,那么与X在体内结合的蛋白质Y也能沉淀下来。
优点:保留蛋白质的修饰和结合状态,同时所需的样本量较少。
缺点:Co-IP特异性较低。
九、噬菌体展示技术(PDT)
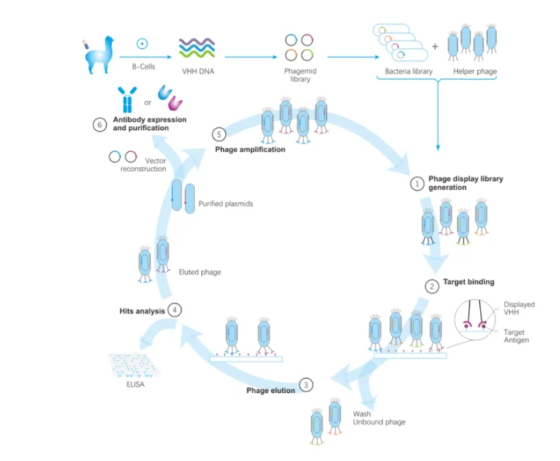
原理:在编码噬菌体外壳蛋白基因上连接一单克隆抗体的DNA序列,当噬菌体生长时,表面就表达出相应的单抗,再将噬菌体过柱,柱上若含目的蛋白,就会与相应抗体特异性结合。
优点:具有简便、高通量的特点,可以直接评价相互结合的特异性。
缺点:不是所有的序列都能在噬菌体中获得很好的表达,因部分蛋白质功能的实现需要折叠、转运、膜插入和络合,导致在体内筛选时需外加选择压力。
十、邻位连接技术(PLA)
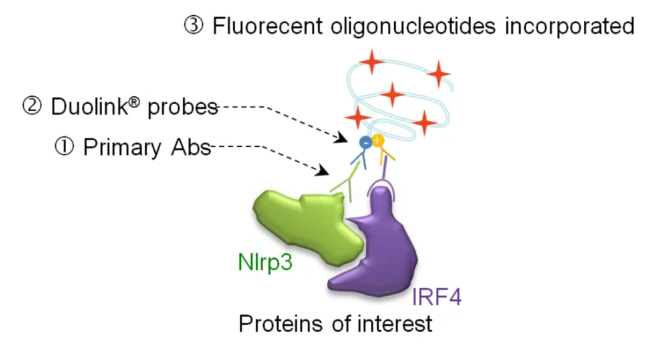
原理:采用核酸适体(aptamer)或单/多克隆抗体-寡聚核苷酸复合物作为邻位连接探针(proximity probes)。当一对邻位探针同时识别同一个目标蛋白分子(一个蛋白复合体中的两个蛋白)时,它们将在空间位置上相互临近,通过连接反应形成可扩增的DNA标签序列,该标签序列能够反映待测蛋白的表达及互作情况。
优点:可识别同一目标的两个不同抗原表位,灵敏度和精确度远高于单个表位识别;通过一对邻位探针环化并且进行滚环扩增,更有利于信号的放大;可以凭借探针锚定目标蛋白,从而准确地定位目标;可以捕获瞬时的相互作用和低丰度蛋白的相互作用。
缺点:暂无一
十一、NanoBiT 蛋白互补技术
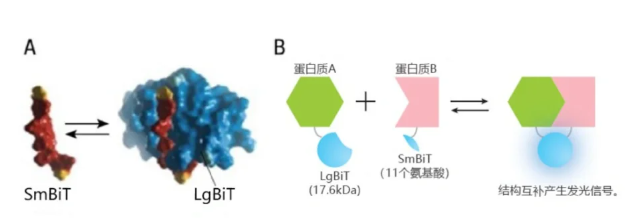
原理:利用两个特定的互补蛋白质片段,在相互作用后形成一个完整的蛋白质复合物。其中,一个蛋白质片段被称为“受体”,另一个蛋白质片段被称为“配体”。当两个互补的蛋白质片段相互接近时,它们会通过非共价相互作用结合在一起,形成一个稳定的复合物。
优点:高度特异性、强大的亲和力以及灵活的应用。
缺点:早期需要摸索LgBiT和SmBiT构建在不同的N端或C端的组合。
十二、抗体与蛋白质阵列技术
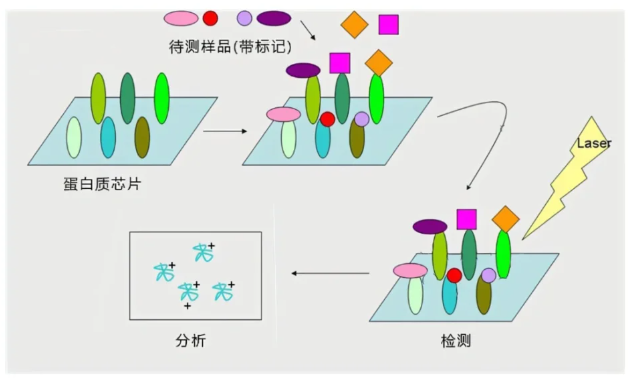
原理:以蛋白质分子作为配基(探针蛋白),将其有序地固定在固相载体(滴定板、滤膜、玻璃片等)的表面形成微阵列;用带有荧光标记的蛋白质(或其它分子)与之作用,经漂洗将未结合的成分洗去,经荧光扫描等监测方式测定芯片上各点的荧光强度。然后通过荧光强度分析蛋白质与蛋白质之间相互作用的关系。
优点:具有微量化、高通量、高灵敏度的特点;样品要求低,可直接对不经处理的各种体液和分泌物进行检测。
缺点:成本过高,需一系列昂贵的精密仪器;芯片的标准化问题。
十三、荧光共振能量转移技术(FRET)
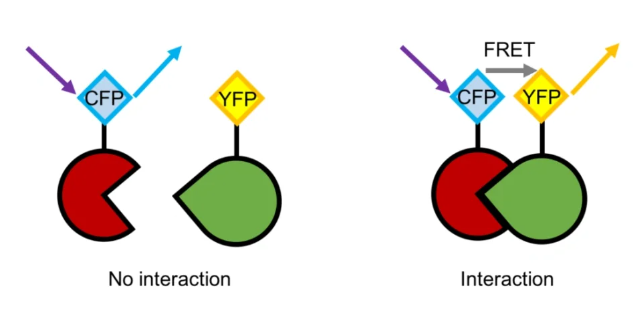
原理:在两个荧光发色基团(如供体荧光基团“CFP”和受体荧光基团“YFP”)在足够靠近时,供体分子吸收一定频率的光子后被激发到更高的电子能态,在该电子回到基态前,通过偶极子相互作用,实现了能量向邻近的受体分子转移。
优点:能检测到瞬时、较弱的蛋白质相互作用,以及俩蛋白的细胞分布和作用位点。
缺点:光谱可能存在重叠,影响实验结果。
十四、双分子荧光互补技术(BIFC)
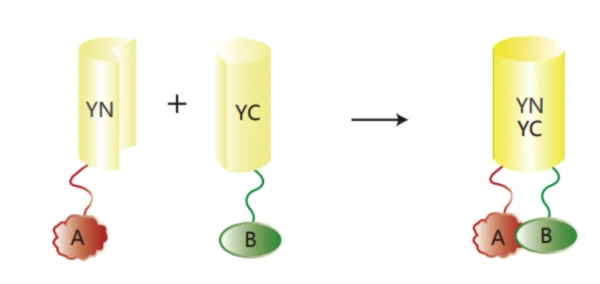
原理:荧光蛋白可从特定的位点被分开,产生两个非荧光活性片段。当两片段分别被融合到相互作用的蛋白上时,会因蛋白的相互作用力而被拉近、发生互补从而重新构建成有活性的荧光蛋白并在激发光下产生荧光。因此可以直接通过观察荧光有无来判断蛋白是否发生相互作用。
优点:能简单方便地通过观察荧光鉴定蛋白质相互作用。
缺点:对温度敏感,不能实时反应蛋白质的结合和分离情况。
十五、生物发光能量共振转移技术(BRET)
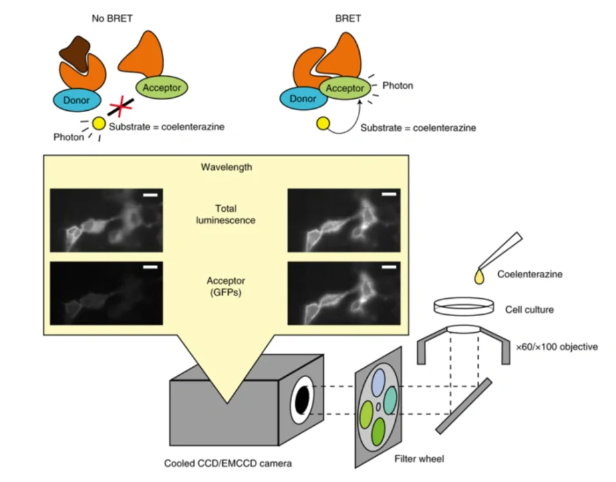
原理:一种流行的基于邻近的检测方法,即将一种目标蛋白与生物发光能量供体融合,其他蛋白质与荧光能量受体融合,可监测活体内的蛋白质-蛋白质相互作用和构象重排细胞。
优点:不依赖于激发光,检测背景低;不需要漂白,细胞损坏更少;同时也避免了自发荧光干扰。
缺点:需要配对的底物较贵。
 文章推荐
文章推荐