
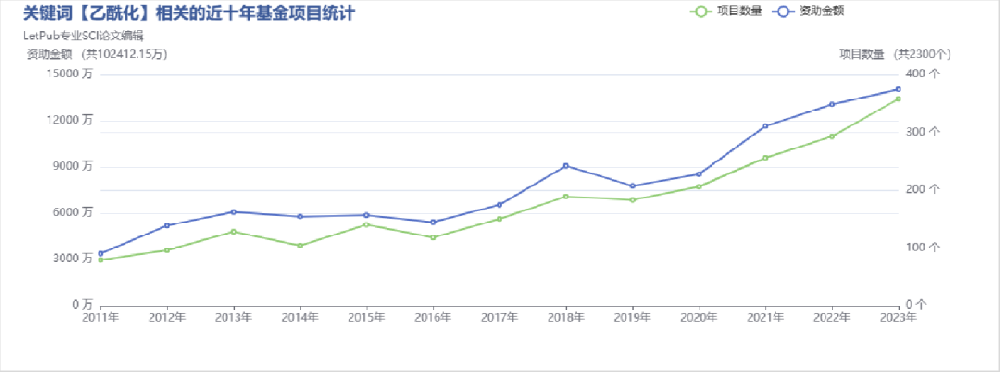
为什么要着重讲解乙酰化呢?我们发现近几年的国家自然科学基金的资助项目中,蛋白质乙酰化修饰的相关项目不断增多。这说明了蛋白质乙酰化修饰是当前生命科学和基础医学研究的热点之一!
一、乙酰化修饰的基础概念
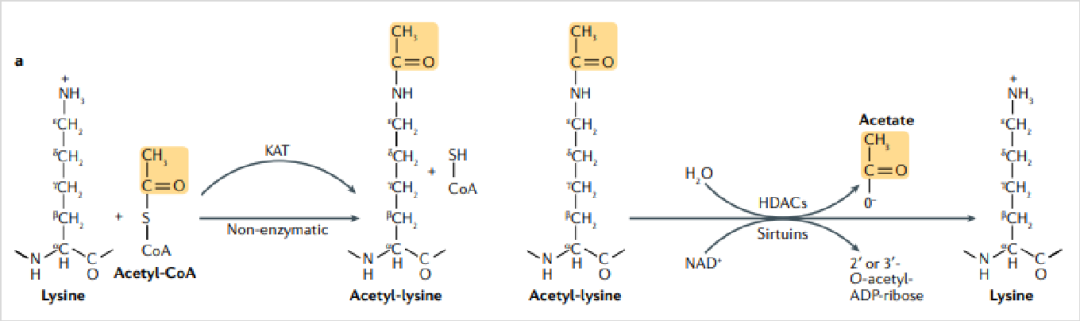
乙酰化修饰是原核和真核生物所共有的一种蛋白翻译后修饰形式,是指在蛋白质原有基础上面嫁接上乙酰化基团,即在乙酰基转移酶(或非酶)的催化下,将乙酰基团转移并添加在蛋白赖氨酸残基或蛋白N端上的过程。
乙酰化的修饰主要分为两类:蛋白N端的乙酰化修饰和蛋白赖氨酸上的乙酰化修饰。其中赖氨酸乙酰化是通过KAT(赖氨酸乙酰化酶)将乙酰基从乙酰辅酶A转移到赖氨酸的ε-氨基侧链而产生的。赖氨酸乙酰化是一种动态的翻译后修饰过程,这个过程是可逆的,在调控蛋白质功能、染色质结构和基因表达中起重要作用。
二、乙酰化修饰的调控酶
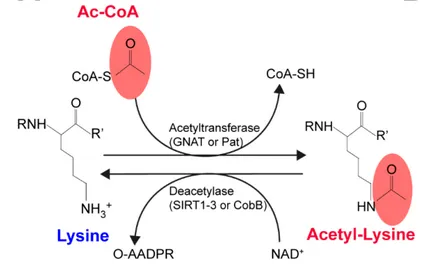
乙酰化修饰的调控酶包括组蛋白乙酰转移酶和去乙酰化酶,两者通过调节组蛋白和转录因子的乙酰化水平,在控制细胞生命活动中发挥着重要作用。
乙酰化酶又称为组蛋白乙酰转移酶,是使组蛋白乙酰基化,减弱组蛋白与DNA的紧密结合能力,促进转录的酶。主要成员有主要成员有Gcn5、PCAF、Elp3、Hatl、Hpa2等;MYST家族;P300/CBP家族等。
去乙酰化酶则是一类蛋白酶,对染色体的结构修饰和基因表达调控发挥着重要的作用。主要成员有HDACl、HDAC2、HDAC3、HDAC4、HDAC5、SirTl-7等。
三、乙酰化修饰如何影响蛋白质的功能
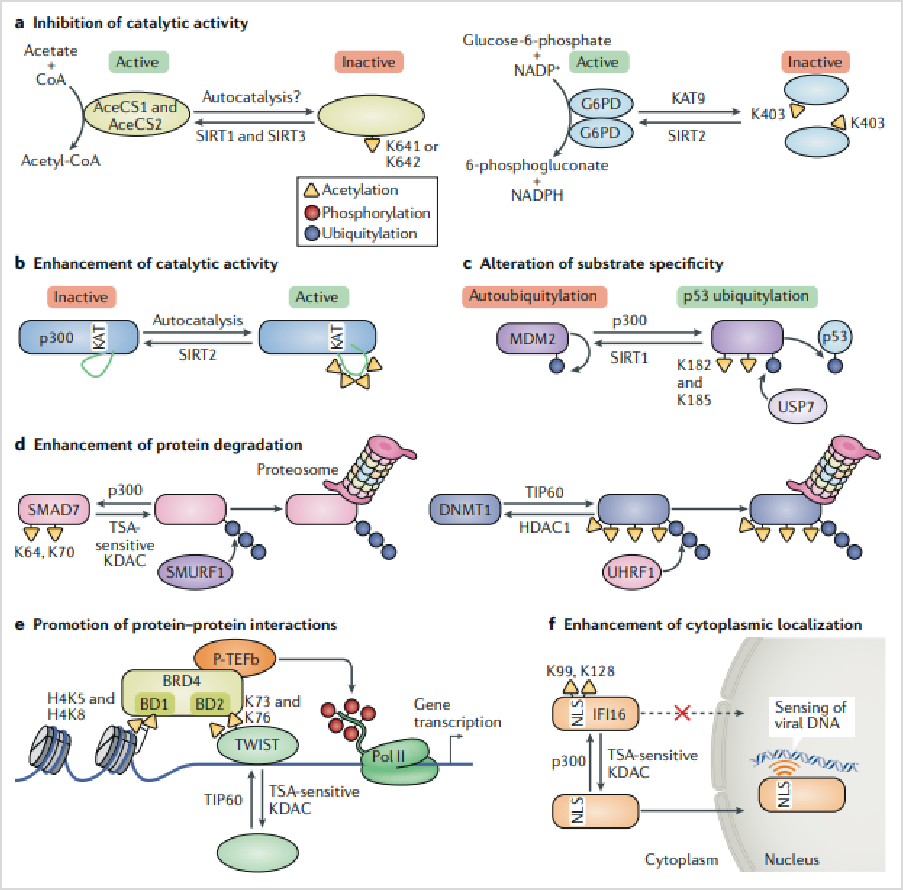
抑制酶的活性。例如:乙酰辅酶A合成酶1和乙酰辅酶A合成酶2分别定位于细胞质基质和线粒体。乙酰化过程会抑制ACSS1和ACSS2的活性,而SIRT1和SIRT3介导的去乙酰化能恢复ACSS1和ACSS2的活性。
增强酶的活性。乙酰化酶可以发生自乙酰化,乙酰化的KATs的活性可以得到提高。这种机制类似于蛋白激酶的自磷酸化。
改变酶-底物特异性。乙酰化能改变E3泛素连接酶MDM2的底物特异性。MDM2的Lys182、Lys185位点会被p300乙酰化,使MDM2与去乙酰化酶USP7结合,从而使MDM2的底物p53泛素化。
蛋白降解的调控。乙酰化既可以调控蛋白酶体依赖的蛋白降解体系,又可以调控不依赖蛋白酶体的蛋白降解体系。
促进蛋白质-蛋白质相互作用。例如转录因子C-ets-1(ETS1)在其氨基末端的两个残基上的乙酰化可增强其与BRD4的相互作用,并促进RNA聚合酶II(PolII)的释放。
调节亚细胞定位。比如病毒感染触发病毒DNA传感器γ干扰素诱导蛋白16(IFI16)NLS的p300依赖性乙酰化,从而促进其细胞质定位。
四、乙酰化修饰的研究思路
下面我们来说说乙酰化修饰的研究思路,一般包括以下五个步骤:
确定目标蛋白是否存在乙酰化;
筛选乙酰化酶/去乙酰化酶;
鉴定乙酰化位点;
解析乙酰化对目标蛋白的影响;
探究目标蛋白乙酰化的生物学功能。
五、乙酰化研究思路的案例分析
接下来通过一篇相关的高分文献(SCFD1的乙酰化调节SNARE复合物的形成和自噬体与溶酶体的融合),带大家了解如何将乙酰化结合到自己的研究课题中。

01目标蛋白是否存在乙酰化
首先作者用两种广谱去乙酰酶抑制剂TSA(HDAC家族抑制剂)和NAM(Sirtuin家族抑制剂)共同处理细胞后,发现SCFD1的乙酰化水平显著增加。由此可以确定SCFD1存在乙酰化修饰。
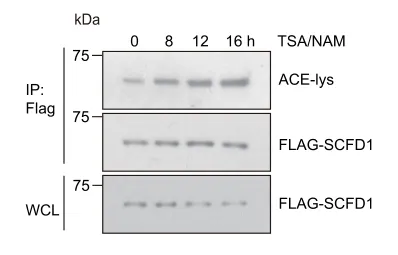
02乙酰化水平受到怎样的调控
作者用MTOR抑制剂和营养缺乏培养基处理细胞后,检测FLAG-SCFD1的乙酰化程度,可以看到FLAG-SCFD1在营养缺乏的条件下明显乙酰化,但这种乙酰化的程度在自噬刺激条件下急剧下降(图A-B)。
接着作者用Torin1在不同时间处理稳定表达FLAG-SCFD1的HEK293T细胞,发现自噬诱导以时间依赖的方式降低了SCFD1的乙酰化水平(图C-D)。
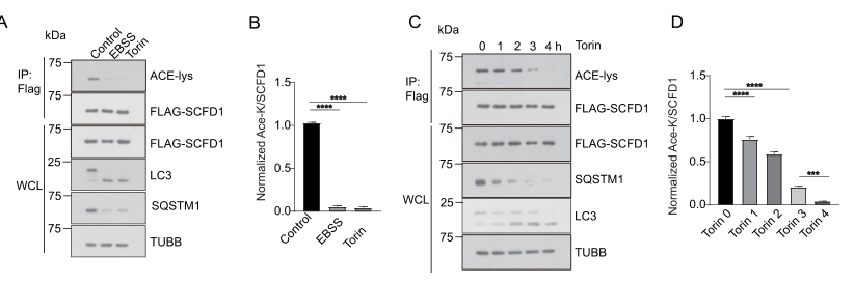
03筛选乙酰化酶/去乙酰化酶
在哺乳动物细胞中,介导蛋白发生乙酰化修饰的主要包括P300、TIP60、PCAF等乙酰化转移酶。一般只需要在细胞内进行简单的筛选,如在细胞内分别高表达或者敲除/低以上乙酰转移酶,检测目标蛋白的乙酰化水平,基本就可以确定目标蛋白的乙酰化由哪个乙酰转移酶介导。
在这里,作者首先选用了四种乙酰转移酶,即CREB、EP300、KAT2B、KAT5/TIP60、与FLAG-SCFD1一起转染HEK293T细胞(图A-B),发现敲除KAT2B而不敲除KAT5会降低SCFD1的乙酰化水平(图C-D)。且用KAT2B特异性抑制剂Garcinol处理过表达FLAG-SCFD1的HEK293T细胞,可显著降低SCFD1乙酰化(图G)。
作者进一步利用体外实验证明SCFD1被KAT2B/PCAF乙酰化(图H)。
我们还可以发现:在表达SCFD1[2KR](双突变体)和SCFD1[3KR](三突变体)的细胞中,KAT2B/PCAF对SCFD1的乙酰化作用减弱(图I)。
由此我们可以得出:KAT2B/PCAF是SCFD1潜在的乙酰化转移酶。
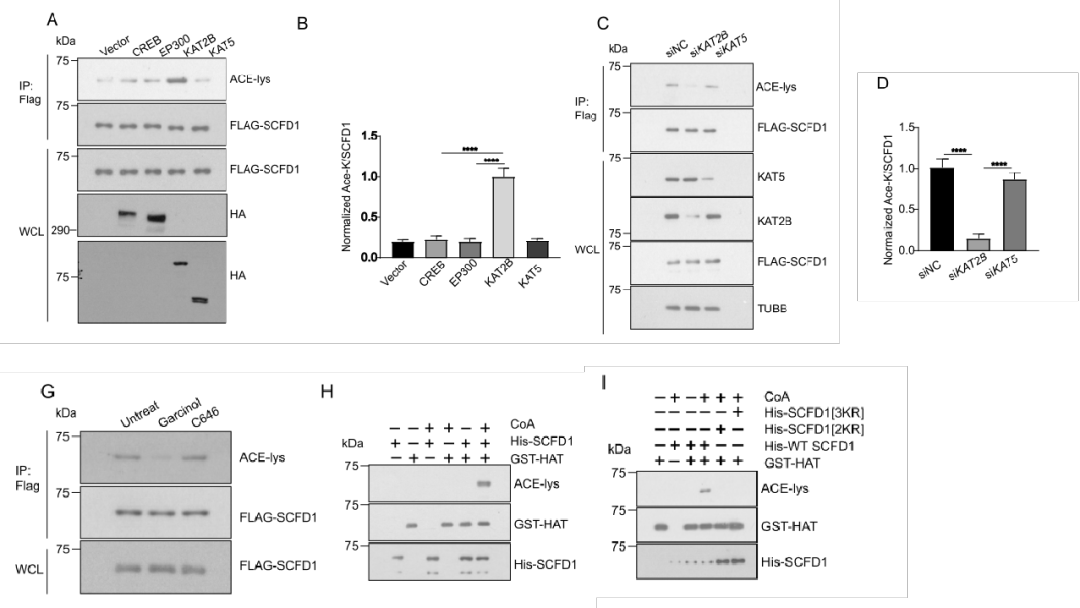
确定乙酰化转移酶后,作者利用两种不同的去乙酰化酶在细胞内作了简单的筛选,以确定去乙酰化酶。
作者用SIRT家族去乙酰酶抑制剂NAM和HDAC家族去乙酰酶抑制剂TSA分别或一起处理细胞,发现SIRT抑制剂NAM对SCFD1乙酰化的影响大于HDAC抑制剂TSA。这一结果表明,SIRT家族成员是SCFD1的潜在去乙酰酶(图A-B)。
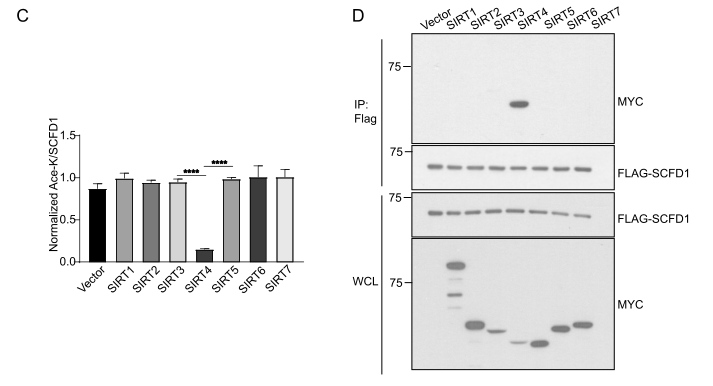
在确定SCFD1的去乙酰化过程由SIRT家族介导后。作者用FLAG-SCFD1和MYC-SIRT1-SIRT7一同转染HEK293T细胞,发现只有SIRT4过表达才能降低SCFD1乙酰化水平(图C-D)。
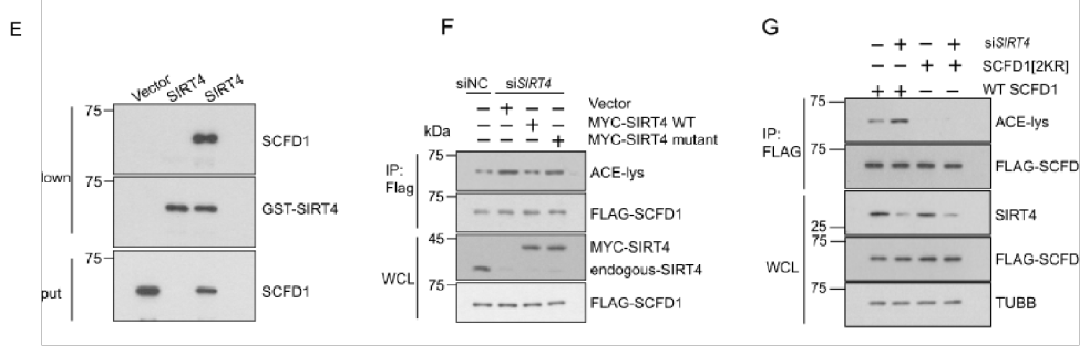
在得知SIRT4与SCFD1的相互作用后(图E),作者使用特定的siRNA敲除SIRT4,发现SCFD1乙酰化水平比过度表达野生型SIRT4的细胞更高(图F)。当SIRT4被敲除时,与野生型SCFD1相比,SCFD1[2KR](突变体)的乙酰化没有进一步增加(图G)。
由此我们可以得出:SIRT4是SCFD1潜在的去乙酰酶。
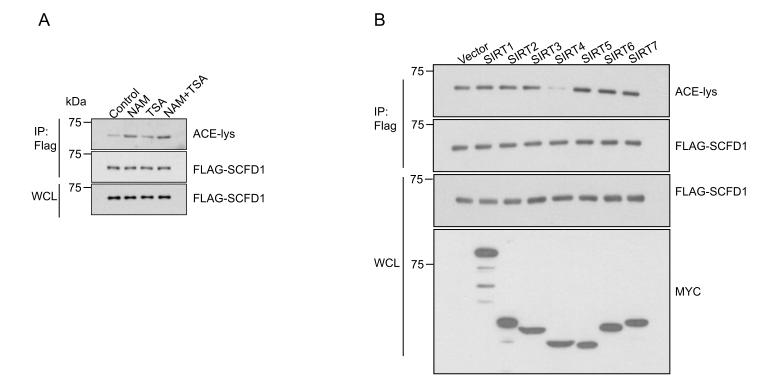
04鉴定乙酰化位点
接下来,作者对SCFD1的乙酰化位点进行了确定:乙酰化主要发生在赖氨酸(K)上。
一般确定乙酰化位点有两种常用的方法,分别是在细胞内直接纯化目标蛋白进行质谱鉴定;或是在体外利用纯化的目标蛋白与乙酰转移酶进行体外乙酰化反应,将反应后的目标蛋白进行质谱鉴定。无论哪种方法确定的乙酰化位点,都要通过构建突变体进行最后的确定。
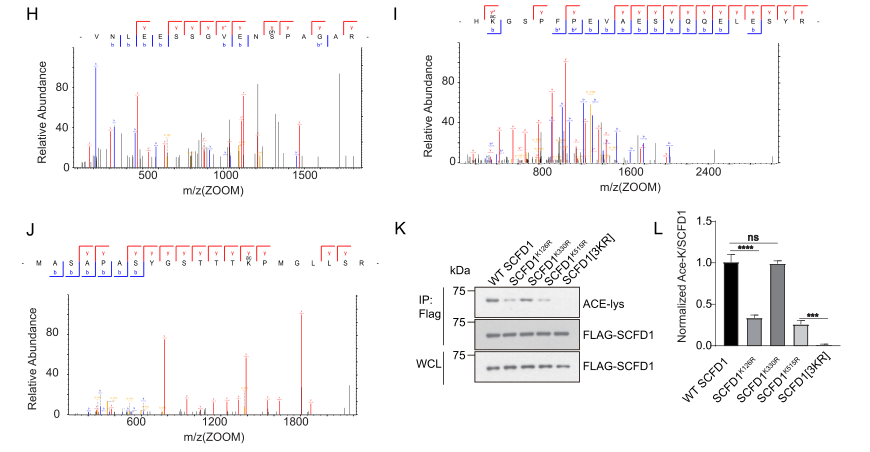
作者首先通过质谱分析鉴定了三个乙酰化位点:K126、K330和K515(图H-J)。
接着作者用WT FLAG-SCFD1和突变体转染HEK293T细胞,以进行位点的验证。与野生型SCFD1和SCFD1(K330R)突变体相比,作者发现SCFD1(K126R)和SCFD1(K515R)突变体显示出更少的SCFD1乙酰化(图K-J)。
根据这一结果,可以得出SCFD1的主要乙酰化残基是K126和K515。
05生物学功能
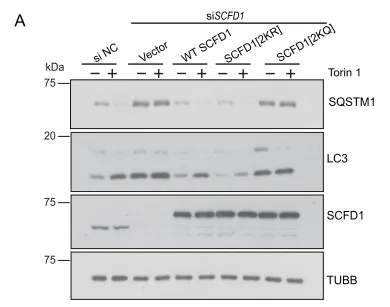
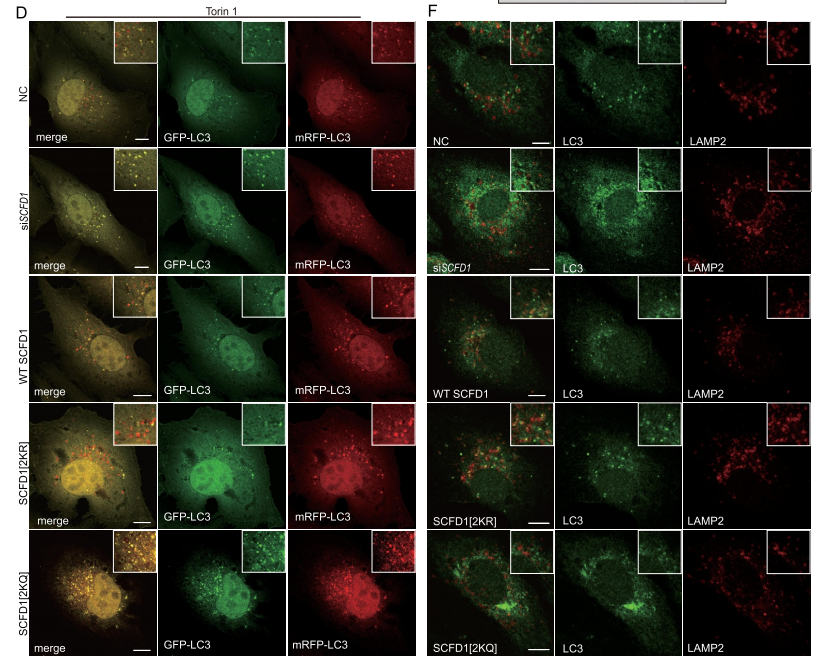
由于p62主要作为自噬受体在自噬过程中发挥作用。因此,作者检测了诱导自噬条件下细胞内泛素化蛋白的降解情况和对细胞活力的影响。在SCFD1敲低的细胞中,LC3和自噬底物SQSTM1/p62的水平显著升高,这些水平在表达SCFD1[2KR](去乙酰化模拟物)的细胞中可以有效地恢复,但在表达SCFD1[2KQ](乙酰化模拟物)的细胞中却不能恢复(图A)。且与WT SCFD1细胞相比,表达SCFD1[2KR]的细胞中自噬体与溶酶体融合的程度更高(图D)。
在表达SCFD1[2KR]突变体的细胞中,内源性LC3(自噬体指标)和LAMP2(溶酶体指标)的共定位高于表达WT SCFD1的细胞(图F)。
由此可以得出SCFD1乙酰化损害自噬通量。
最后我们总结回顾一下文章的整体脉络。
首先,作者确定了在SCFD1存在乙酰化修饰,随后针对乙酰化修饰的调控机制进行研究,发现营养缺乏和自噬诱导共同调控SCFD1的乙酰化水平,接着作者进一步筛选确定了SCFD1的潜在乙酰化酶KAT2B/PCAF,潜在去乙酰化酶SIRT4,以及乙酰化位点K126和K515,最后,作者探究了SCFD1乙酰化的生物学功能,即损害自噬通量。总的来说,这一研究揭示了SCFD1的乙酰化修饰可调节自噬体成熟。
 文章推荐
文章推荐