
在生物信息领域,PCA、t-SNE和UMAP是三种重要的降维与可视化方法,此前我们了解了PCA图的具体解读方法,今天我们就来了解一下UMAP图具体应该怎么看吧~
一、【UMAP图】定义&用途
1.定义:UMAP图是一种基于非线性降维的可视化方式,将高维数据映射到二维或三维空间,并保持数据之间的相对距离和结构,从而使得聚类、异质性和样本间的差异更为明显。
2.用途:
(1)单细胞数据分析:UMAP图可以用于单细胞测序数据的降维分析,帮助研究人员探索细胞的状态和细胞间的相似性,从而揭示细胞分化和功能编排的机制。
(2)基因组学和转录组学:UMAP图可以帮助分析高维转录组数据,主要揭示基因库表达与特定生物过程的关联,有助于理解复杂的生物学功能网络。
(3)生物标志物发现:通过UMAP分析高通量数据,可以发现潜在的生物标志物,从而诊断疾病、监测治疗效果以及开发新的治疗方法。
二、【UMAP图】案例解读
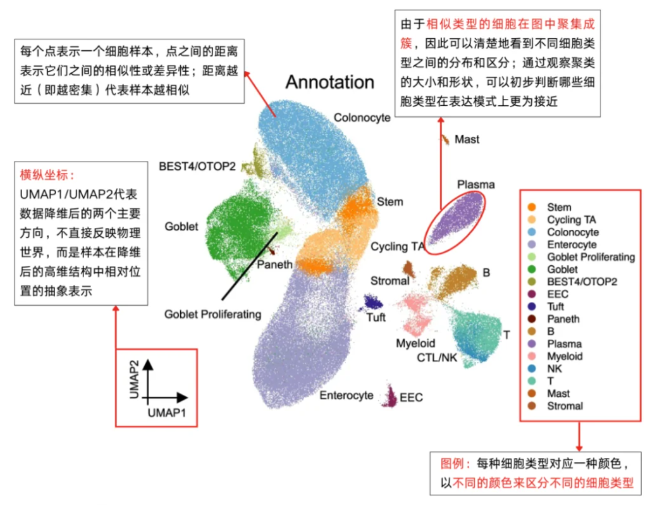
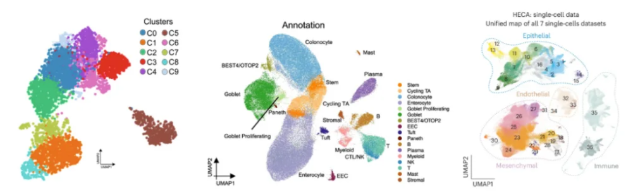
该图是内窥镜样本中155093个细胞的UMAP,揭示了上皮室中的主要细胞类型为enterocytes/colonocytes(肠细胞/结肠炎细胞)、stem cells(干细胞)、goblet cells(杯状细胞)、goblet proliferating cells(杯状增殖细胞)、BEST4/OTOP2细胞、tuft cells(簇状细胞)、EEC(肠内分泌细胞)、Paneth细胞等;非上皮室中的主要类型为T细胞、B细胞、plasma(浆细胞)、myeloid cells(髓细胞)、CTL/NK(硬脊膜杀伤细胞)、mastcells(肥大细胞)和stromalcells(基质细胞)等。
三、相关疑问&解答
1.UMAP、PCA和t-SNE三者之间的异同点?
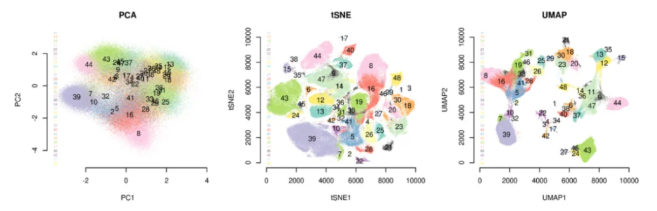
(1)线性与非线性:PCA属于线性方法,适合处理线性可分的数据;而t-SNE和UMAP则属于非线性方法,更适合于处理复杂的非线性结构数据。例如在转录组学中PCA可以用于探索基因之间的线性关系,而t-SNE和UMAP则主要揭示基因表达模式的非线性结构和细胞间的复杂关系。
(2)计算复杂度:PCA的计算相对简单且快速,而t-SNE和UMAP的计算复杂度较高。尤其在处理大型数据集时,t-SNE可能会变得非常耗时。在处理大规模的单细胞转录组数据时,UMAP通常比t-SNE更高效。
(3)可视化效果:PCA通常只能显示数据的线性结构,而t-SNE和UMAP则能够更好地展示数据的非线性结构。其中,PCA分群效果最差,UMAP和t-SNE可以将与相似细胞群相对应的簇聚集在一起。但是与t-SNE相比,UMAP还保留了更多的全局结构,特别是细胞子集的连续性。
(4)参数敏感性:PCA的结果不易受参数设置的影响,而t-SNE和UMAP的结果可能易受到初始化参数的影响。不同的参数设置可能会得到不同的可视化效果,并且在生物信息学中,参数敏感性会影响结果的可重复性。
2.UMAP如何保证降维后数据的结构信息的保留?
(1)局部邻域保留:UMAP计算每个点的局部邻域,然后将这些邻域嵌套在超平面中。这种嵌套保持了原数据中的局部结构。
(2)相似性度量:UMAP使用一种非线性变换来保持邻近点之间的相似度,可以捕捉数据中的复杂结构。
(3)维度嵌入:UMAP在低维空间中对点进行重排,使得低维空间的距离大致对应原数据中的相似性。
 文章推荐
文章推荐